Die verblüffenden Eigenschaften der Coronavirus-Hülle
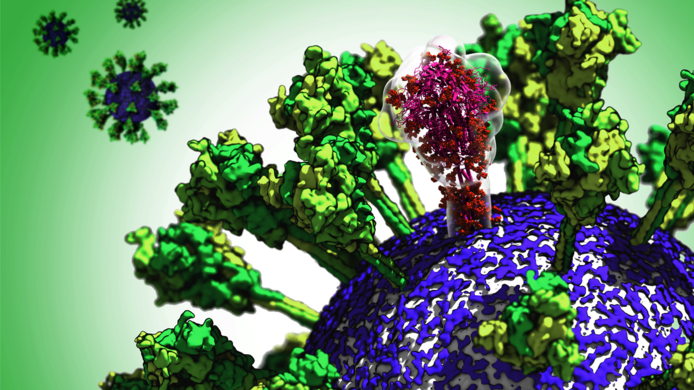
FWF: Herr Sikora, Sie absolvieren als Erwin-Schrödinger-Stipendiat einen Forschungsaufenthalt am Max-Planck-Institut für Biophysik in Frankfurt. Was war ursprünglich der Gegenstand Ihres Projekts? Mateusz Sikora: Ich untersuchte, wie lebende Zellen aneinandergebunden sind. Es gibt bestimmte Proteine der Zellmembranen, die wie Haken sind und die Zellen aneinanderheften. Dieser Prozess ist nicht gut verstanden und es ist nicht klar, wie er ausgelöst wird, also warum manche Arten der Bindung sehr stark sind und andere nicht. Als dann die Pandemie begann, sah ich schnell, dass ich genau die richtigen Werkzeuge hatte, um die Anbindung des Coronavirus an die Zelle zu untersuchen, denn auch Viren nützen spezielle Proteine, um sich an die Wirtszelle zu heften – eine Voraussetzung für das Infizieren der Zelle. FWF: Wie veränderte sich ihr Fokus durch die Pandemie? Sikora: Ich erinnere mich, dass wir hier am Institut im Februar, als die Pandemie Italien erfasste, auf dem Flur ins Gespräch kamen, ob etwas über die Oberfläche des Virus bekannt war. Wir dachten, dass wir vielleicht helfen könnten, und ich begann mit einigen anderen Leuten, Professor Gerhard Hummer und meinen Kollegen Sören von Bülow, Florian Blanc, Michael Gecht und Roberto Covino, eine Taskforce zum Coronavirus zu organisieren. Die Oberfläche ist wichtig, weil sie der einzige Teil des Virus ist, der nach außen hin sichtbar ist und damit der einzige Anknüpfungspunkt für das Immunsystem oder für Medikamente. Es gibt ein Oberflächenprotein, das wir Spike nennen, und das grob die Form eines Lollipops hat. Damit heftet sich das Virus an die Wirtszelle und leitet die Verbindung der Zellmembran des Virus mit jener der Zelle ein.
„Für die Simulationen nutzten wir alles an Wissen, das wir über das ältere Coronavirus SARS-CoV-1 finden konnten. “
Wir fanden, dass es bereits Forschungsgruppen gelungen war, Teile der Struktur des Proteins zu entschlüsseln. Sein Oberteil war bereits in guter Auflösung bekannt, doch nicht, wie es mit dem Virus verbunden ist. Man wusste außerdem nicht, wie dieses Bild mit der Wirklichkeit „in vivo“ übereinstimmt. Vor diesem Hintergrund begannen wir, ein Modell des Proteins zu entwickeln und nutzten alles an Wissen, das wir über das ältere Coronavirus SARS-CoV-1 finden konnten. Für diese Simulationen bekamen wir 20 Millionen Stunden Rechenzeit am Supercomputer SuperMUC in Deutschland zugesprochen. Zudem bekam ich vom FWF eine Verlängerung meines Stipendiums genehmigt, speziell im Zusammenhang mit Covid-19, und konnte mich so vier weitere Monate voll dieser Arbeit widmen.

FWF: Sie arbeiteten also mit Computersimulationen. Doch das Projekt nutzte auch Elektronenmikroskopie-Bilder. Sikora: Richtig, das war eine schöne Synergie. Zur selben Zeit als wir die Modelle entwickelten, hatte die Gruppe um Martin Beck hier am Institut gerade Proben des Coronavirus aus München bekommen und sie für mikroskopische Untersuchung vorbereitet. Sie arbeiteten unabhängig von uns, bis sie begannen, mehrere Bilder zu einem einzigen hochauflösenden Bild des Spike-Proteins zu kombinieren. Sie stellten fest, dass sie das Verbindungsstück des Proteins mit dem Virus nicht auflösen konnten. Sie wandten sich dann an uns und wir wussten bereits, dass diese Verbindung sehr beweglich und elastisch war. Wir konnten also erklären, warum sie diese Struktur in ihren Bildern nicht sehen konnten. FWF: Was ist so ungewöhnlich an dieser Verbindung? Sikora: Als wir mit unseren Modellrechnungen begannen, sahen wir schnell, dass der Kopf des Spikes sehr stabil war, aber dass die Verbindung zum Virus über mehrere Gelenke verfügte, wo sie sich biegen kann. Es gab ein Gelenk, das wie ein Knie funktionierte, eines, das wie ein Sprunggelenk war und eines wie eine Hüfte. Wir nannten es dann „Bein“, weil die Analogie so stark war. FWF: Was bedeutet das für die Funktion des Proteins? Sikora: Wenn Sie sich vorstellen, dass das Virus relativ klein ist im Vergleich zur Wirtszelle, diese ist fast tausendmal so groß, dann ist die Oberfläche der Wirtszelle für das Virus praktisch eine flache Oberfläche. Wäre der Spike nur einfach eine feste Spitze auf der Virusoberfläche, könnte nur ein Protein an die Wirtszelle anbinden. Die Idee ist, dass das Coronavirus flexible Beine hat und so mehrere Proteine auf einmal die Wirtszelle berühren. Das erhöht die Chancen, dass es tatsächlich zu einer Verbindung kommt.
„Ich hatte das Glück, dass ich die Werkzeuge für diese Arbeit bereits im Rahmen meines Schrödinger-Stipendiums entwickelt hatte.“
FWF: Welche Konsequenzen hat das für die Forschung, speziell für die Entwicklung von Impfstoffen? Sikora: Wir wurden bereits von verschiedenen Unternehmen und Forschungsgruppen kontaktiert. Die gute Nachricht für sie ist, dass die reale Situation in vivo jener sehr ähnlich ist, die sie in vitro in den Labors hatten. Sie können dem vertrauen, was sie bisher über die Struktur des Virus wussten. Abgesehen davon könnte das Bein des Spike-Proteins ein guter Anknüpfungspunkt für ein Medikament oder einen Impfstoff sein. Wenn man einen Antikörper fände, der an das Bein bindet, wäre es für das Virus vermutlich sehr schwer, so zu mutieren, dass es das Anbinden verhindert. Auch zwischen SARS-Cov-2 und dessen Vorgänger hat sich das Bein kaum verändert. Es scheint sich also um einen wichtigen Teil für die Funktion des Virus zu handeln. FWF: Ihre Ergebnisse erregten international große Aufmerksamkeit. Was bedeutet diese Entdeckung für Sie als junger Forscher? Sikora: Ich hatte das Glück, dass ich die Werkzeuge für diese Arbeit bereits im Rahmen meines Schrödinger-Stipendiums entwickelt hatte und dass ich die Verlängerung genehmigt bekam. Für mich ist es jetzt wichtig, mich nicht zurückzulehnen. Wir bereiten gerade mehrere neue Publikationen vor. Eine davon dreht sich um Ergebnisse einer großen Simulation, mit der wir unter anderem untersuchten, welche Teile des Spike-Proteins sogenannte Epitope sind, die Anknüpfungspunkte für das Immunsystem bilden und auch als Anhaltspunkte für die Entwicklung von Impfstoffen benutzt werden können.
Mateusz Sikora ist Physiker am Max-Planck-Institut für Biophysik in Frankfurt, wo er einen Forschungsaufenthalt im Rahmen eines Erwin-Schrödinger-Stipendiums des Wissenschaftsfonds FWF absolviert. Zuvor war er Postdoc am Institute of Science and Technology (IST) Austria. Er interessiert sich für die Modellierung von biologischen Molekülen, insbesondere die Interaktion von Proteinen, die in biologischen Membranen eingebettet sind.
Internationale Mobilität
Die Erwin-Schrödinger-Stipendien sind ein Programm des Wissenschaftsfonds FWF, das sich an junge Forschende in Österreich wendet, die nach dem Doktorat Auslandserfahrung sammeln wollen. Sie bekommen dabei die Möglichkeit, neue Fachgebiete und Methoden kennenzulernen, wobei eine Rückkehr nach Österreich finanziell unterstützt wird. Die Dauer ist auf zehn Monate bis zwei Jahre ausgelegt.
Publikation




